运动想象脑电CSP-SVM支持向量机二分类
本文基于一个公开数据集和一个BCI竞赛数据集,提供一个SVM-CSP运动想象二分类demo。公开数据集来自论文” Open Access Dataset for EEG+NIRS Single-Trail Classification”,BCI竞赛数据集来自2008年BCI Competition中Data sets 2b数据集。
软件环境及数据背景
MATLAB2016a + pycharm-community-2020.2,Python 3.8.3 + scikit-learn 0.23.1,matlab处理数据格式为mat,python处理数据格式为npy。
公开数据集
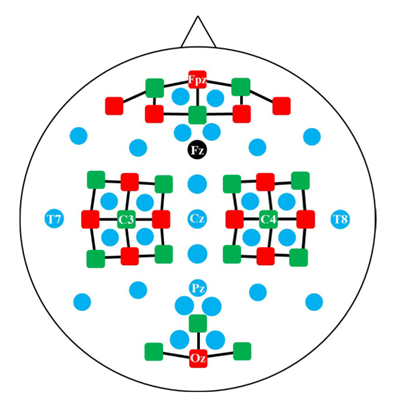
论文” Open Access Dataset for EEG+NIRS Single-Trail Classification”提供了一个使用脑电图(EEG)和近红外光谱(NIRS)的混合脑机接口(BCIs)开放存取数据集,本文只使用其中的脑电数据。有29名健康参与者(14名男性和15名女性,年龄为28.5±3.7岁)参与了数据采集,脑电采样频率为1000Hz,作者提供的数据集已经降采样到200Hz。图1位电极位置。
本文选用’FCC5h’、’FCC6h’、’FCC3h’、’FCC4h’、’Cz’、’CCP5h’、’CCP6h’、’CCP3h’、’CCP4h’ 9个通道,即图1中C3和C4周围8个通道加Cz通道。
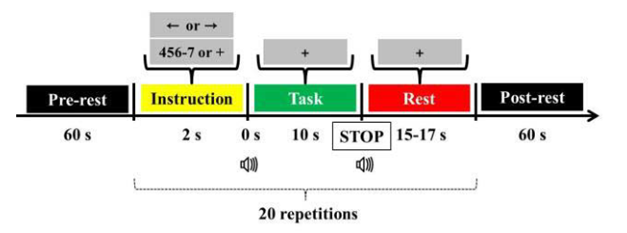
图二为左右运动想象的实验范式。
每一阶段包括1分钟的实验前休息期、20次重复给定任务和1分钟的实验后休息期。任务开始时用2秒的时间对任务进行视觉介绍,然后用10秒的时间进行任务和休息时间(从15到17秒随机分配)。详细介绍请查阅该论文。
BCI竞赛数据集
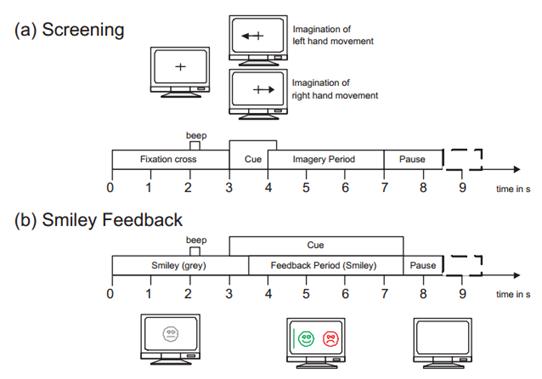
08年竞赛数据在论文中引用很多了,都比较熟悉,图3为这批数据的范式。
其中(a)对应的是01T和02T系列无反馈数据,(b)对应的是03T、04E和05E系列有反馈数据。提供数据被试有9人,每人有以上五组数据,共45组数据。记录电极为C3、C4和Cz三个通道,采样频率为250Hz,数据集下载链接为http://www.bbci.de/competition/iv/,这批数据本文将全部使用。
正文
为了方便叙述,下文将论文公开数据集记为DATA1,BCI竞赛数据集记为DATA2。根据以往论文,在数据使用之前,对数据进行了8~30Hz带通滤波,保留运动想象诱发的ERD /ERS现象主要集中区,mu节律(8~13 Hz) 和beta节律(14~30 Hz),滤波器使用matlab提供的切比雪夫Ⅱ型滤波器,在通带内单调下降,阻带内等波纹,过度带宽较小。代码如下:
Wp = [8 30]/200*2;
Ws = [6 33]/200*2;
Rp = 3;
Rs = 30;
[n, Ws] = cheb2ord(Wp, Ws, Rp, Rs);
[b,a] = cheby2(n,Rs,Ws);
epo.imag = proc_filtfilt(epo.imag, b, a);epo是DATA1合成脑电连续数据后的结构体,epo.imag是脑电运动想象数据结构体,proc_filtfilt函数由BBCI工具箱提供。使用前需要下载工具箱并添加MATLAB路径。
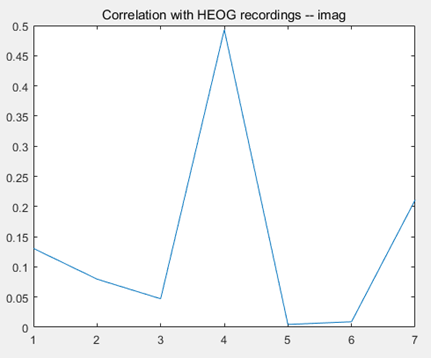
对DATA1和DATA2两批数据均只做了滤波处理后进行使用了,也尝试使用了去基线、重定义参考和去眼电等预处理操作,但分类结果表明不如只做滤波处理的效果好,尤其自动去眼电操作,因为eeglab去眼电需要凭借经验且千样本处理耗时。作者使用fastica算法将epo.imag.x数据进行独立成分提取并与DATA1中提供的两个通道眼电数据做相关度比较,去除相关度最高的两个成分。
为了不过分影响数据量,设定独立成分小于7个时,不进行去除操作,但结果仍是差了8个百分点,故只保留滤波操作。DATA2预处理与DATA1相同。
滤波后直接对数据进行CSP特征提取操作,不清楚的地方可以对照” CSP-Designing optimal spatial filters for single-trial EEG classification in a movement task”论文来看,比如程序中说的”协方差矩阵”在文中的定义,其维度即脑电通道数,还有数据格式和处理顺序导致的微小变化等。CSP特征提取函数如下:
function [ F, csp_feature, csp_label ] = CspFeature( inputData, inputLabel, featDim )
% -----------------------------------
% 求取输入数据的CSP特征
% -----------------------------------
% Output:
% F -- 空间滤波器
% csp_feature -- inputData提取的CSP特征,(采样点数,样本数)
% csp_label -- csp_feature对应的标签
% Input:
% inputData -- 输入数据,(采样点数,通道数,样本数)
% inputLabel -- inputData对应的标签,0/left,1/right
% featDim -- 提取特征对数
channels = size(inputData, 2);
trials = size(inputData, 3);
CovData = zeros(channels, channels, trials);
for i = 1:trials
sigData = inputData(:,:,i);
sigDataX = sigData'*sigData;
CovData(:,:,i) = sigDataX/trace(sigDataX);
end
C_left = sum(CovData(:, :, inputLabel == 0), 3);
C_right = sum(CovData(:, :, inputLabel == 1), 3);
C = C_left + C_right;
[Vc, Dc] = eig(C);
P = diag(diag(Dc).^(-1/2))*Vc';
S_left = P*C_left*P';
[Vs, Ds] = eig(S_left);
[~, index] = sort(diag(Ds), 'descend');
Vs = Vs(:, index);
F = P'*Vs;
F = F(:, [1:featDim channels-featDim+1:channels]);
data_left = inputData(:, :, inputLabel == 0);
data_right = inputData(:, :, inputLabel == 1);
left_num = size(data_left ,3);
right_num = size(data_right ,3);
csp_left = zeros(featDim*2, left_num);
csp_right = zeros(featDim*2, right_num);
for i=1:left_num
csp_left(:, i) = log(var(data_left(:, :, i)*F));
end
for i=1:right_num
csp_right(:, i) = log(var(data_right(:, :, i)*F));
end
csp_feature = [csp_left, csp_right];
csp_label = [zeros(1, left_num), ones(1, right_num)];
end输入输出参数都已经写好了注释并标注了数据格式,使用前需要确认输入数据没有NAN和全为0的样本,比如DATA2就需要对NAN进行处理。
写好CSP特征提取函数之后,合并DATA1中29个被试运动想象脑电数据,根据范式截取运动想象全部10秒的数据,为了对比DATA2将featDim设定为3,该值不能超过通道数。
得到29个被试一共1740个CSP样本,训练集与测试集之比为3:1,使用机器学习库scikit-learn提供的支持向量机算法,主要代码如下:
ss = StandardScaler()
x_train = ss.fit_transform(x_train)
x_test = ss.fit_transform(x_test)
linSvc = LinearSVC()
linSvc.fit(x_train, y_train)
y_predict = linSvc.predict(x_test)
print('The Accuracy of Linear SVC is', linSvc.score(x_test, y_test))
print('precision&recall&f1-score of Linear SVC:', '\n',
classification_report(y_test, y_predict,
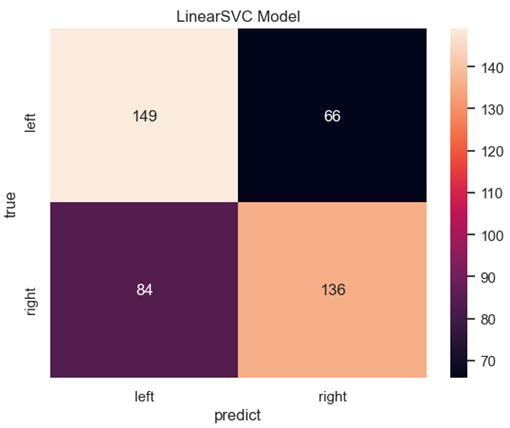
target_names=['左', '右']))运行得到SVM-CSP-DATA1分类准确率为65.51%,对应的混淆矩阵如图5所示。
输出:
| The Accuracy of Linear SVC is 0.6551724137931034 precision&recall&f1-score of Linear SVC: precision recall f1-score support 左 0.64 0.69 0.67 215 右 0.67 0.62 0.64 220 accuracy 0.66 435 macro avg 0.66 0.66 0.65 435 weighted avg 0.66 0.66 0.65 435 |
对于DATA2数据,9个被试,每个被试5组数据。但下载得到的是gdf格式,根据提供的数据说明文档” BCI Competition 2008 – Graz data set B”可知,可以下载biosig工具箱,将gdf格式转换为matlab处理的mat格式,需要注意的是不能使用matlab设置路径来添加biosig,这样会报错添加失败,其实在biosig>INSTALL文件中已经写了操作方法,在biosig文件夹下运行install.m文件,不要清空工作区,紧接着就可以运行加载语句了:
[BCI2bData, HDR]=sload(gdfName_list{id});程序中BCI2bData是脑电数据,HDR是相关信息结构体,如HDR.TRIG记录着样本数,HDR.EVENT中记录着打标点等参数,作者这里直接使用样本数结合采样率来划分样本的,根据范式提取了3~7秒数据,没有使用打标点。
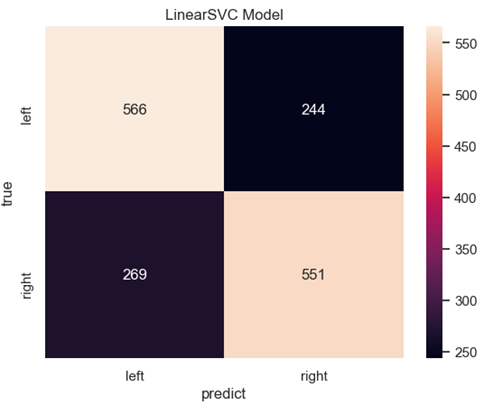
合并所有mat数据后提取CSP特征得到6520个样本,同样的训练集与测试集之比为3:1,运行分类程序得到SVM-CSP-DATA2分类准确率为68.52%,对应的混淆矩阵如图6所示。
输出:
| The Accuracy of Linear SVC is 0.6852760736196319 precision&recall&f1-score of Linear SVC: precision recall f1-score support 左 0.68 0.70 0.69 810 右 0.69 0.67 0.68 820 accuracy 0.69 1630 macro avg 0.69 0.69 0.69 1630 weighted avg 0.69 0.69 0.69 1630 |
综上SVM对两个数据集CSP特征的分类准确率分别为0.65和0.68,文末提供了资源下载连接。关于代码使用DATA1进行演示;关于数据DATA1提供了被试01的原始数据以供参考,DATA2提供了被试01的第一组数据以供参考;关于工具箱文中使用到的bbci和biosig工具箱也已包含。
另关于其功率谱特征使用CNN卷积神经网络分类demo,可阅读姊妹篇”运动想象脑电PSD-CNN卷积神经网络二分类”。
或扫码搜索关注微信公众号“燕山徐一”,后台回复“运动想象CSP”获取代码。